Coding-Party 基于飞桨的农作物智能识别系统
基于深度学习的病虫害检测方法不仅具有重要的学术研究价值,而且具有非常广阔的市场应用前景。其次轻量化模型的构建需要成为未来研究考虑的重要组成部分,使得农作物病害智能识别可以
项目背景
联合国粮食及农业组织最近的一份报告表明,每年农业生产的自然损失中有三分之一以上是由农业病虫害造成的。需要考虑的农业病虫害众多,依赖于实验室观察和实验的传统方法很容易导致错误的诊断。为加快转变农业发展方式,农业部组织开展农作物病虫害专业化统防统治与绿色防控融合推进,逐步实现农作物病虫害全程绿色防控的规模化实施、规范化作业。融合推进可以有效提升病虫害防治的组织化程度和科学化水平,是实现病虫综合治理、农药减量控害的重要内容,也是转变农业发展方式、实现提质增效的重大举措。在保障防治效果的同时,农产品质量符合食品安全国家标准,生态环境及生物多样性有所改善。

项目目的
农作物只要是出现病虫害问题,不仅仅会给人们的生活和环境产生非常严重的影响,也会给农民的收入造成不利的影响。
在广大农村地区大部分农民都会首选传统农药方式来进行病虫害防治:
-
对于病虫害防治意识比较差;
-
应用传统农药防治不符合实际情况,如药量不合理、周期不合理等。不但没有有效的防治,而且给农作物造成比较大的伤害,同时也出现严重的环境污染的问题。最终就造成了农作物病害防治的效果极差,使得农作物无法正常的生长。到了最后人工防治难度增大、成本提升,后果严重。
-
为加快转变农业发展方式,农业部组织开展农作物病虫专业化统防统治与绿色防控融合推进,逐步实现农作物病虫害全程绿色防控的规模化实施、规范化作业。融合推进可以有效提升病虫害防治的组织化程度和科学化水平,是实现病虫综合治理、农药减量控害的重要内容,也是转变农业发展方式、实现提质增效的重大举措。在保障防治效果的同时,农产品质量符合食品安全国家标准,生态环境及生物多样性有所改善。
应用深度学习对农作物进行准确的病害识别并推荐合适的防治措施,创造出能为农作物看病的“植物医生”,一定程度上起到综合防治的效果和标准,以更好地消除农作物病害,促进农作物健康的生长,保证农作物的产量。
技术路线图
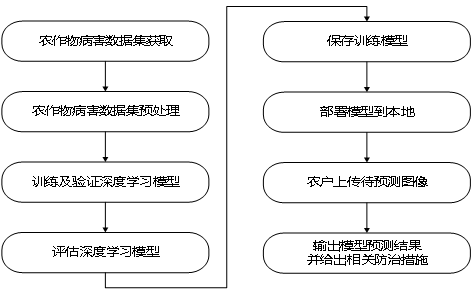
系统架构图
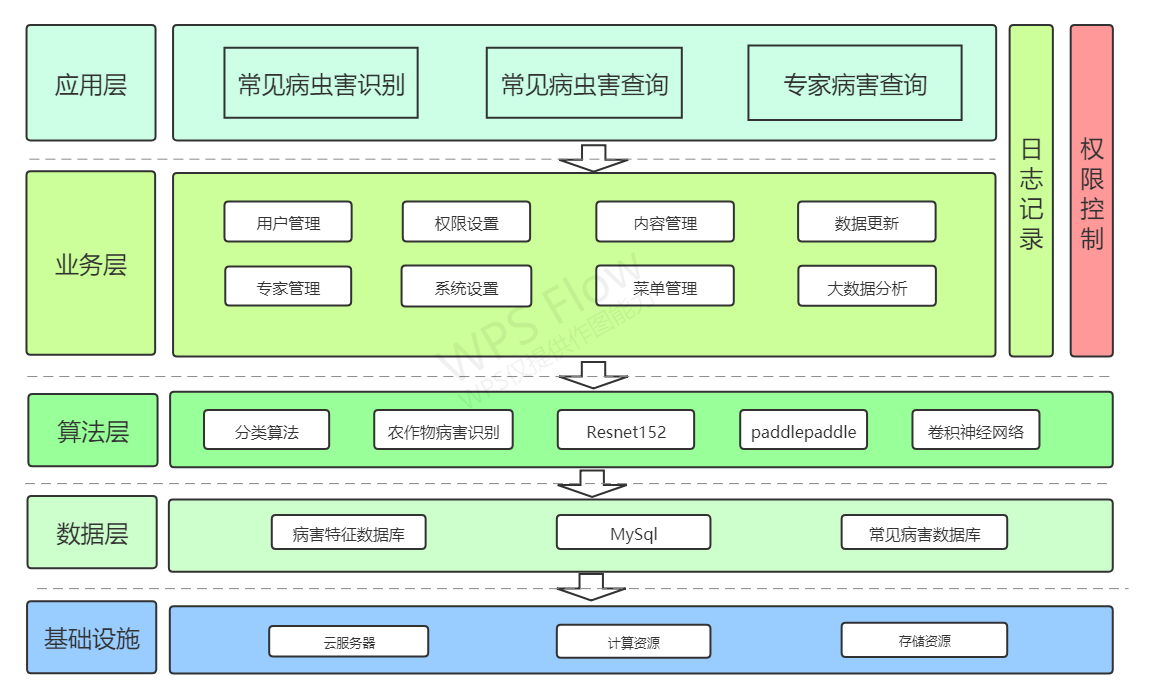
项目创新点
本作品将基于AI Challenger农作物叶子图像数据集包含10种植物的27种病害,合计61个分类(按“物种-病害-程度”分)的特性同时结合实地采集的数据,主要从以下几个方面考虑改进:
- ①增加一定的农作物种类与细分粒度
绝大多数能识别出来的病虫害,等能识别出来的时候已经太晚了。比如草莓白粉病,发病初期症状极为不明显(非常薄的白色绒毛状斑点),只有大量白色粉末状真菌长满叶片(或者果实)的时候才能很明显的看到。 - ②优选模型,注重预测精度,增加考虑因素
存在一部分病虫害在发生危害之前根别看不见病/虫原,如植物根部的线虫,藏于叶子后的蚜虫等情况,将监控不局限于图像的分析识别。 - ③追溯预测其患病因素
针对目前世面上的系统只做了简单的识别功能,缺少对病虫害产生原因的分析,本项目将完善与改进这一方向
数据集
本研究基于AI Challenger农作物叶子图像数据集包含10种植物(苹果、樱桃、葡萄、柑桔、桃、草莓、番茄、辣椒、玉米、马铃薯)的27种病害(其中24个病害有分一般和严重两种程度),合计61个分类(按“物种-病害-程度”分)的特性,训练图像总数为31718张,测试图像总数为4540张。每张图包含一片农作物的叶子,叶子占据图片主要位置。
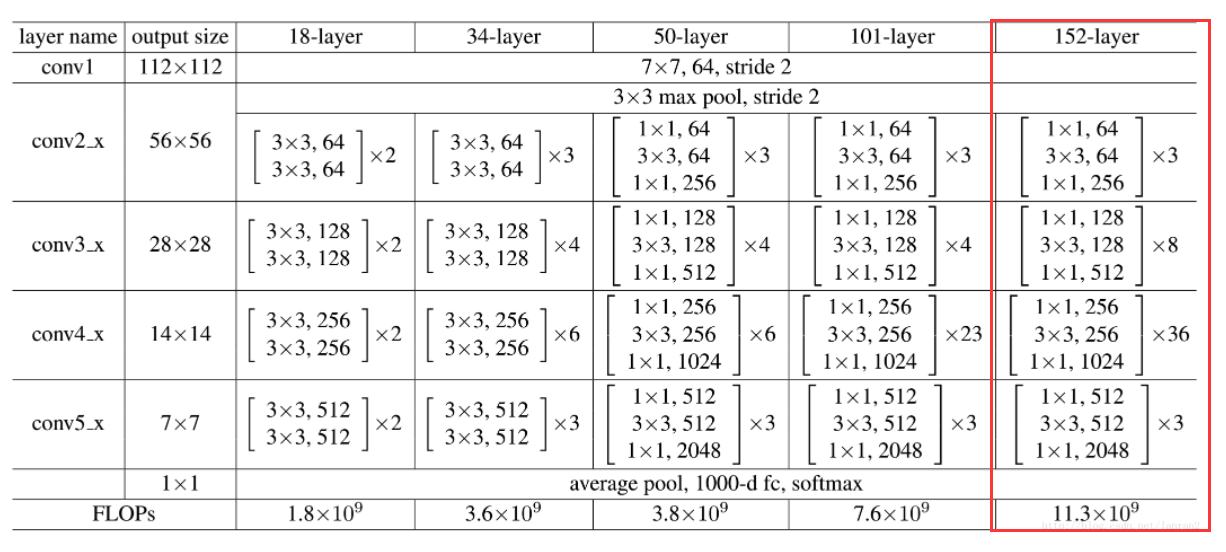
网络模型
ResNet通过改变学习目标,即不再学习完整的输出F(x),而是学习残差H(x)−x,解决了传统卷积层或全连接层在进行信息传递时存在的丢失、损耗等问题。通过直接将信息从输入绕道传输到输出,一定程度上保护了信息的完整性。同时,由于学习的目标是残差,简化了学习的难度。
解压数据集
#!unzip /home/aistudio/data/data101323/data.zip
数据预处理
import paddle
import paddle.nn.functional as F
import numpy as np
import cv2
import json
import math
import random
import os
from paddle.io import Dataset # 导入Datasrt库
filename = "AgriculturalDisease_trainingset/AgriculturalDisease_train_annotations.json"
f_open = open(filename)
fileJson = json.load(f_open)
train_data = []
for i in range(len(fileJson)):
img1=cv2.imread("AgriculturalDisease_trainingset/images/"+fileJson[i]['image_id'])
img2=cv2.resize(img1, (128,128), interpolation=cv2.INTER_AREA)/255
r=[]
g=[]
b=[]
r.append(img2[:, :, 0])
g.append(img2[:, :, 1])
b.append(img2[:, :, 2])
one_data = np.concatenate((r,g,b),axis=0)
one_data = paddle.to_tensor(one_data,dtype="float32")
train_data.append([one_data,fileJson[i]['disease_class']])
filename = "AgriculturalDisease_validationset/AgriculturalDisease_validation_annotations.json"
f_open = open(filename)
fileJson1 = json.load(f_open)
test_data = []
for i in range(len(fileJson1)):
img1=cv2.imread("AgriculturalDisease_validationset/images/"+fileJson1[i]['image_id'])
img2=cv2.resize(img1, (128,128), interpolation=cv2.INTER_AREA)/255
r=[]
g=[]
b=[]
r.append(img2[:, :, 0])
g.append(img2[:, :, 1])
b.append(img2[:, :, 2])
one_data = np.concatenate((r,g,b),axis=0)
one_data = paddle.to_tensor(one_data,dtype="float32")
test_data.append([one_data,fileJson1[i]['disease_class']])
/opt/conda/envs/python35-paddle120-env/lib/python3.7/site-packages/paddle/fluid/layers/utils.py:26: DeprecationWarning: `np.int` is a deprecated alias for the builtin `int`. To silence this warning, use `int` by itself. Doing this will not modify any behavior and is safe. When replacing `np.int`, you may wish to use e.g. `np.int64` or `np.int32` to specify the precision. If you wish to review your current use, check the release note link for additional information.
Deprecated in NumPy 1.20; for more details and guidance: https://numpy.org/devdocs/release/1.20.0-notes.html#deprecations
def convert_to_list(value, n, name, dtype=np.int):
数据读取
from paddle.static import InputSpec
import paddle.nn.functional as F
print("-------end readData--------")
class MyDataset(Dataset):
"""
步骤一:继承paddle.io.Dataset类
"""
def __init__(self, mode='train'):
"""
步骤二:实现构造函数,定义数据读取方式,划分训练和测试数据集
"""
super(MyDataset, self).__init__()
if mode == 'train':
self.data = train_data
else:
self.data = test_data
def __getitem__(self, index):
"""
步骤三:实现__getitem__方法,定义指定index时如何获取数据,并返回单条数据(训练数据,对应的标签)
"""
data = self.data[index][0]
label = self.data[index][1]
return data, label
def __len__(self):
"""
步骤四:实现__len__方法,返回数据集总数目
"""
return len(self.data)
# s_tra_data,s_tra_label = split_data(train_data,train_label,batch_size=32)
# s_tes_data,s_tes_label = split_data(test_data,test_label,batch_size=32)
#数据读取
train_loader = paddle.io.DataLoader(MyDataset("train"), batch_size=16, shuffle=True)
test_loader = paddle.io.DataLoader(MyDataset("test"), batch_size=16, shuffle=True)
-------end readData--------
定义训练过程
epoch_num = 20 #训练轮数
batch_size = 16
learning_rate = 0.0001 #学习率
val_acc_history = []
val_loss_history = []
def train(model):
print('start training ... ')
# turn into training mode
model.train()
opt = paddle.optimizer.Adam(learning_rate=learning_rate,
parameters=model.parameters())
for epoch in range(epoch_num):
acc_train = []
for batch_id, data in enumerate(train_loader()):
x_data = data[0]
y_data = paddle.to_tensor(data[1],dtype="int64")
y_data = paddle.unsqueeze(y_data, 1)
logits = model(x_data)
loss = F.cross_entropy(logits, y_data)
acc = paddle.metric.accuracy(logits, y_data)
acc_train.append(acc.numpy())
if batch_id % 200 == 0 and batch_id != 0:
print("epoch: {}, batch_id: {}, loss is: {}".format(epoch, batch_id, loss.numpy()))
avg_acc = np.mean(acc_train)
print("[train] accuracy: {}".format(avg_acc))
loss.backward()
opt.step()
opt.clear_grad()
# evaluate model after one epoch
model.eval()
accuracies = []
losses = []
for batch_id, data in enumerate(test_loader()):
x_data = data[0]
y_data = paddle.to_tensor(data[1],dtype="int64")
y_data = paddle.unsqueeze(y_data, 1)
logits = model(x_data)
loss = F.cross_entropy(logits, y_data)
acc = paddle.metric.accuracy(logits, y_data)
accuracies.append(acc.numpy())
losses.append(loss.numpy())
avg_acc, avg_loss = np.mean(accuracies), np.mean(losses)
print("[test] accuracy/loss: {}/{}".format(avg_acc, avg_loss))
val_acc_history.append(avg_acc)
val_loss_history.append(avg_loss)
model.train()
开始训练
model = paddle.vision.models.resnet152(pretrained=True,num_classes=61)
train(model)
path = "save_model"
model.train()
开始训练
model = paddle.vision.models.resnet152(pretrained=True,num_classes=61)
train(model)
path = "save_model"
paddle.jit.save(model, path,input_spec=[InputSpec(shape=[16,3,128,128], dtype='float32')])
作品前端实现
前端功能结构图

系统主界面

病虫查询界面

病害识别结果

常见病害查询

更多推荐
 已为社区贡献1436条内容
已为社区贡献1436条内容









所有评论(0)